
Proteomik
Dr. Julian Langer
Unsere Methoden
Siehe hier für Impressionen
Wir beteiligen uns auch an externen Kooperationen und sind derzeit Mitglied im SPP 2002 (Small proteins in prokaryotes) der DFG und im MOEL-SOEL-Programm des BMBF.
Aktuelle Forschungs-Highlights
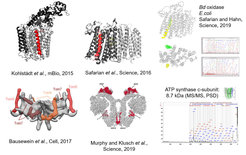
Direkte Sequenzierung zur Identifizierung neuer Untereinheiten von Membranproteinkomplexen.
Membranproteinkomplexe stellen anspruchsvolle Ziele für die Bottom-up-Proteomik dar. Insbesondere kleine, in die Membran eingebettete Untereinheiten haben oft keine proteolytischen Spaltstellen oder ionisieren nicht effizient in ESI. Zusammen mit Bruker Daltonics haben wir ein rapifleX MALDI-TOF/TOF-Massenspektrometer modifiziert, das nun die Aufnahme von informationsreichen PSD-MS/MS-Spektren bis zu m/z 9000 ermöglicht. Wir haben diese Technik bereits erfolgreich eingesetzt, um mehrere neue Untereinheiten in Membranproteinkomplexen zu identifizieren (Kohlstädt et al., mBio, 2015; Safarian et al., Science, 2016; Bausewein et al., Cell, 2017; Murphy et al., Science, 2019; Hahn and Safarian et al., Science, 2019).
HDX-MS von Membranproteinkomplexen.
Wir haben erfolgreich Ligandenbindungsstellen und aktivitätsassoziierte Konformationsdynamik in mehreren Membranproteinkomplexen charakterisiert, darunter ein Natrium:Proton-Antiporter (Eisinger et al., PNAS, 2017), ein MATE-Transporter (Eisinger et al., JMB, 2018) und ein Molybdat-Speicherprotein (Brünle et al., PNAS, 2019). Wir erweitern diese Technik derzeit auf große Membranproteinkomplexe.
Proteom-Profiling.
Wir haben erfolgreich das Proteom-Remodeling bei homöostatischer synaptischer Plastizität in primären hippocampalen Neuronen charakterisiert, indem wir die BONCAT-Technologie nutzten (Schanzenbächer et al., Neuron, 2016; Schanzenbächer et al., eLife, 2018). Wir haben auch den Proteom-Umsatz in diesem System überwacht (Dörrbaum et al., eLife, 2018) und untersuchen derzeit seine Rolle bei der homöostatischen Skalierung.
Wir haben einen Arbeitsablauf etabliert, der die Analyse von zelltypspezifischen Proteomen aus lebenden Tieren ermöglicht, und haben die Proteomdynamik in Tieren untersucht, die einer angereicherten Umgebung ausgesetzt waren (Alvarez-Castelao et al., Nature Biotechnology, 2017). Eine detaillierte Beschreibung der Methode finden Sie unter Alvarez-Castelao et al., Nature Protocols, 2019. Kürzlich haben wir erfolgreich die Proteinsynthese in neuronalen Prozessen untersucht (Biever and Glock et al., Science, 2020).
MS-Imaging.
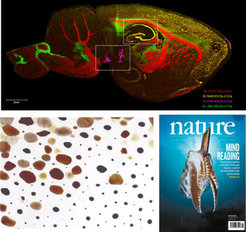
Wir haben erfolgreich die molekularen Komponenten in Sepia Chromatophoren charakterisiert, die eine Schlüsselrolle in ihrem Tarnsystem spielen. Mit einer Kombination aus UPLC-UV-ESI-MS/MS, MS-Imaging und direkter Infusions-UHR-MS identifizierten wir Xanthomatin in diesen Chromatophoren, ein redox-schaltbares Molekül, das bereits zuvor beschrieben wurde und eine Rolle bei der Reifung und Färbung von Libellen spielt (Reiter et al., Nature, 2018).
Instrumente
Wir arbeiten mit acht Instrumenten von Bruker, Thermo und Waters, darunter...
Hier finden Sie Eindrücke
Die Systeme sind an nano-UPLCs (Thermo/Dionex Ultimate3000 und easy-1200; Bruker nanoElute) und analytische UPLCs (Thermo/Dionex Ultimate3000; Waters Acquity) gekoppelt.

